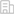大家應該都有過因為身體不適去醫院進行血常規檢查的經歷,這有助于醫生判斷我們的病因并制定相應的治療計劃。當細菌感染上呼吸道時,我們會發現血液中的中性粒細胞比例上升,這是因為激活的中性粒細胞扮演著“免疫警察”的角色,幫助我們的身體清除入侵的細菌。
然而,目前醫院的血常規檢查只能告訴我們中性粒細胞的數量增加了,但不能詳細地告訴我們這些中性粒細胞中有多少是處于活化狀態。事實上,在我們體內有許多不同類型的細胞,它們的狀態會在不同的生理或病理條件下出現動態變化。那么,當面對復雜的實體組織疾病,比如惡性腫瘤,有什么方法可以知道組織中哪些細胞發生了狀態的改變?它們改變的比例又有多高呢?
北京時間2023年7月13日23點,Nature Computational Science在線發表了西湖大學博士研究生宋立陽領導的一項研究,題為“Mixed model-based deconvolution of cell-state abundances (MeDuSA) along a one-dimensional trajectory”。
文章截圖:

原文鏈接:
https://www.nature.com/articles/s43588-023-00487-2
該研究開發了一款名為MeDuSA的方法用以估計組織水平轉錄組數據中處于不同狀態的細胞的豐度。研究團隊將該方法應用于人類癌癥和新冠肺炎等多種疾病,成功地鑒定出了與疾病發生、發展、預后和藥物治療反應相關的細胞狀態,從而促進了我們對不同生物學過程中的細胞狀態動力學的理解。
隨著單細胞轉錄組測序技術(scRNA-seq)的快速發展,我們現在可以高通量地獲取組織中處于不同狀態的細胞的轉錄組。通過結合計算分析手段,我們可以利用單細胞轉錄組信息推斷單個細胞的狀態,從而探究疾病中細胞狀態的變化。
然而,由于單細胞技術的高成本和對生物樣品的高要求,將scRNA-seq技術廣泛應用于大規模生物數據庫仍具有挑戰性。與scRNA-seq相比,組織水平轉錄組測序(bulk RNA-seq)具有成本低、對組織質量要求較低的優點,更適合用于大規模生物數據庫。
然而,因為bulk RNA-seq喪失了單細胞水平的信息,我們無法直接通過轉錄組觀察細胞狀態的變化。那么,我們是否能結合這兩種測序技術的優點,利用小樣本的scRNA-seq作為參考系,然后基于bulk RNA-seq去推斷細胞狀態的轉移,從而在大規模生物數據庫的尺度上研究細胞功能狀態與疾病的關系呢?
本研究開發的MeDuSA方法正是基于這樣的細胞去卷積(cellular deconvolution)策略。它可以利用來自小樣本的scRNA-seq數據作為參考系,去推斷bulk RNA-seq中處于不同狀態的細胞的豐度。過去的細胞去卷積方法由于模型架構的限制,通常只能推斷出細胞類型(如中性粒細胞)的豐度,而無法準確推斷細胞狀態(如活化的中性粒細胞)的豐度。
MeDuSA方法利用混合線性模型擬合單個細胞的轉錄組,系統性地降低了模型殘差,并優化了由于細胞狀態之間強相關性造成的估值偏差,從而能夠精準地估計細胞狀態的豐度(圖1)。
這種結合scRNA-seq和bulk RNA-seq的分析方法突破了在大規模生物數據庫中難以研究細胞狀態的瓶頸,為揭示疾病背后的細胞狀態動力學及其相關的生物學機制邁出了重要一步。
研究人員利用MeDuSA對來自710位捐獻者的食道組織的bulk RNA-seq數據進行了深入分析。他們發現,相較于正常的食道組織,食道腫瘤患者的組織中的上皮細胞明顯富集于黏膜基層(生發層),這一發現與以往的組織學認知相吻合,即食道腫瘤主要起源于食管基層。
研究人員還分析了來自215位捐獻者的外周血數據,結果揭示,與健康捐獻者相比,新冠肺炎(COVID-19)患者血液中的CD8+ T細胞在激活狀態的比例顯著提高,并且這種激活狀態的比例與新冠肺炎的感染程度呈正相關。在對507位黑色素瘤患者的數據分析中,研究人員發現這些患者的腫瘤組織內的CD8+ T細胞大多處于耗竭狀態,并且耗竭狀態的比例與組織中的T細胞受體的擴增程度成正相關。
值得注意的是,他們還發現,CD8+ T細胞的耗竭狀態與黑色素瘤患者的生存時間及對免疫治療的應答效果存在強烈的相關性,這提示了CD8+ T細胞的狀態可能是一個重要的生物指標,對于臨床決定是否使用免疫治療具有重要意義。
此外,通過結合全基因組測序數據,研究人員鑒定出162個基因具有上皮細胞分化依賴性的表達數量性狀位點(eQTL),這表明細胞狀態在遺傳調控中扮演了重要角色。
這項研究還展望了未來的研究方向,提出隨著空間轉錄組學(Spatial transcriptomics)的普及,如何利用空間轉錄組作為參照,從bulk RNA-seq數據中推斷出空間細胞狀態的分布將成為我們需要解決的問題。這一研究方向將進一步深化我們對疾病背后的細胞狀態動力學的理解,為未來疾病的預防和治療開啟更多的可能性。
MeDuSA方法已經被整合為一個用戶友好的分析軟件,并已在以下鏈接開源:
https://github.com/LeonSong1995/MeDuSA。
本研究的第一作者是西湖大學2021級博士研究生宋立陽,西湖大學楊劍教授是本文的通訊作者。該研究得到了國家自然科學基金、西湖實驗室、西湖教育基金會以及西湖大學未來產業研究中心和高性能計算中心的支持。
團隊簡介
楊劍,西湖大學教授,2003年本科畢業于浙江大學,2008年于浙大取得博士學位,同年赴澳大利亞昆士蘭醫學研究所從事博士后研究工作。2012年加入澳大利亞昆士蘭大學,歷任研究員、高級研究員、副教授、教授。2020年加入西湖大學生命科學學院。主要致力于統計遺傳學、基因組學研究,以及人類復雜性狀和疾病(如:身高、肥胖、精神分裂和癌癥)的大數據分析。
楊劍團隊工作獲得了國家自然科學基金委、浙江省自然科學基金委、西湖大學和西湖實驗室的經費支持。團隊誠邀對統計遺傳學或系統與合成生物學感興趣的有志青年加盟,歡迎聯系[email protected]。