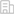抗生素的發現是20世紀醫學史上最輝煌的成就之一。然而,隨著抗生素大量和無序使用,抗生素耐藥問題則已成為21世紀人類不得不面對的嚴峻挑戰。抗生素耐藥問題不僅是困擾臨床的一大難題,“超級耐藥細菌”的不斷出現更是引起公眾的極大恐慌,成為全球普遍關注的公共生物安全問題。從遺傳學角度,耐藥性的產生是細菌為應對環境變化,尤其是抗生素選擇壓力下進化的產物。在進化歷程中,抗生素耐藥基因的每個核苷酸位點都可能發生突變,有些突變使細菌對抗生素更敏感,有些會導致更耐藥。在抗生素選擇壓力下,更耐藥的突變類型會更容易被篩選出來,并引起逐步擴散和傳播。
2022年4月29日,中山大學中山醫學院陳小舒教授、楊建榮教授和田國寶教授合作研究團隊在Molecular Biology and Evolution在線發表題為“Prediction of Antibiotic Resistance Evolution by Growth Measurement of All Proximal Mutants of Beta-Lactamase”的研究論文。該工作以臨床廣泛流行的超廣譜β-內酰胺酶耐藥基因blaCTX-M為研究模型,構建了包含blaCTX-M所有單點突變基因型的大腸桿菌克隆文庫,結合高通量測序和神經網絡模型實現了對耐藥基因進化的溯源和前瞻性預測。
研究團隊利用混雜引物、以Fusion PCR的方法在耐藥基因中引入突變,構建包含所有單堿基突變的等位基因庫,同時每個基因型均帶有隨機的條形碼。為了確保突變基因型在每個細胞中拷貝數一致,作者建立了一套高效的整合系統,將突變基因型整合到大腸桿菌MG1655的attHK022位點,最終獲得了等位基因突變體庫,并通過三代測序技術獲得基因型與條形碼的對應關系。結合藥物篩選和二代測序技術,測定每個基因型在不同藥物濃度下的相對生長值(Relative growth),并繪制了相應的適應度景觀(Fitness landscape)。有意思的是,部分位點,如P167、L169、E166,發生突變將顯著提高大腸桿菌的相對生長值和MIC值,而這些突變體的主要富集在超廣譜β-內酰胺酶CTX-M的活性中心(圖1)。

圖1. 抗生素耐藥基因進化適應度景觀的建立
研究團隊進一步以適應度景觀為基礎,分別建立了前瞻性預測模型(PEARP)和回顧性溯源模型(PEARR)。PEARP模型可用于預測新的突變基因型是否耐藥,其AUC為0.928。模型預測為耐藥的基因型(包括單點、雙點、三點、四點突變基因型)顯著富集于臨床分離到的基因型。PEARR模型可用于回顧性預測耐藥基因的進化軌跡,且其預測的進化軌跡在SSWM(Strong-Selection-Weak-Mutation)模型下與獨立的藥敏實驗的結果相符(圖2)。

圖2:抗生素耐藥基因進化的回顧性溯源和前瞻性預測模型
綜上所述,研究團隊開發的高通量平臺可用于精準預測抗生素耐藥基因的進化軌跡、輔助抗菌藥物設計和指導藥物使用,為臨床抗生素耐藥性防控提供有力支撐。
中山大學中山醫學院副研究員馮思源、博士研究生吳卓幸、梁婉菲為論文共同第一作者,陳小舒教授、楊建榮教授和田國寶教授為論文通訊作者。